Нейросетевая обработка данных в ГИС GRASS и R
Совместного использования ГИС GRASS и статистического пакета R.
Данная статья продолжает публикацию Анализ данных с использованием GRASS GIS и R, посвященную теме совместного использования ГИС GRASS и статистического пакета R.
Обсудить в форуме Комментариев — 2
В данной статье рассматривается пример использования нейросетевого пакета AMORE и анализ данных в ГИС GRASS.
Оглавление
1. Описание задачи
Процесс нейросетевого анализа данных GRASS GIS рассмотрим на примере конкретной задачи:
Описание задачи: даны снимки одной и той же территории, сделанные в разное время. Предполагается, что некоторая (в процентном соотношении небольшая) часть территории за время, прошедшее между съемками, подверглась изменению. Нужно определить участки изменений.
Ход решения: берем пару последовательных снимков и строим регрессию - по первому снимку предсказываем значения второго снимка. Условие того, что изменению подверглась незначительная часть территории позволяет надеяться, что:
- предсказание второго снимка по первому может быть довольно точным
- те участки, для которых заметно значительное расхождение предсказанного и реального значений и есть изменившиеся участки.
Регрессию будем строить при помощи нейронных сетей. Данные, предназначенные для анализа хранятся в проекте GRASS, используемые снимки - MODIS (используются два канала - (1) красный и (2) ближний инфракрасный). Рассматриваются две даты: 3 апреля 2000 года и 12 июля 2000 года и территория заповедника Черные Земли.
 Композитное RGB изображение 1-2-1, 3 апреля 2000, данные MODIS
Композитное RGB изображение 1-2-1, 3 апреля 2000, данные MODIS
 Композитное RGB изображение 1-2-1, 12 июля 2000, данные MODIS
Композитное RGB изображение 1-2-1, 12 июля 2000, данные MODIS
Т.о. действовать будем следующим образом:
- Предобработка данных: производится в GRASS.
- Построение нейронной сети: производится в R.
- Постобработка данных и анализ результатов: производится в GRASS.
2. Реализация
Предобработка
Будем строить многослойный перцептрон, который берет на входе значения первого и второго каналов снимков от 3-го апреля, а на выходе возвращает предполагаемые значения первого и второго каналов для снимков от 12-го июля. Функцию активации нейронов возьмем сигмоидального типа:
f(u) = 1/(1+ exp(-u)).
Перед построением сети необходимо предварительно пронормировать данные. Выходные значения должны быть нормированы к диапазону изменения функции активации (0, 1), входные же значения нормировать хотя и не обязательно, но, тем не менее, очень желательно.
Конкретный вид нормирующей функции становится ясен после анализа гистограммы распределения входных и выходных данных. В качестве примера возьмем данные по второму каналу каждого снимка:
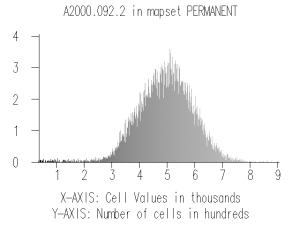 Гистограмма для второго канала (3 апреля)
Гистограмма для второго канала (3 апреля)
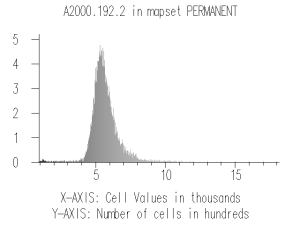 Гистограмма для второго канала (12 июля)
Гистограмма для второго канала (12 июля)
Как видим, первая гистограмма близка к нормальной, поэтому воспользуемся линейной нормировкой с использованием статистических характеристик. Более конкретно, пронормируем данные соответсвующего снимка по формуле:
X = (x - m)/s
Здесь X - нормированное значение, x - исходное значение, m - среднее значение, s - стандартное отклонение.
Со вторым снимком дело обстоит немного сложнее: на гистограмме явно видны длинные хвосты слева и справа, соответственно, линейное нормирование к диапазону (0, 1) приведет к тому, что почти все данные будут сгруппированы в небольшом интервале. Чтобы избежать этого воспользуемся нормировкой при помощи функции активации:
X = f[(x - m)/s]
Здесь X - нормированное значение, x - исходное значение, m - среднее значение, s - стандартное отклонение, f - функция активации нейронов.
С первым каналом дело обстоит аналогично.
Реализация нормирования в GRASS GIS:
- Посмотрим статистику по первому каналу для снимка от 3-го апреля:
r.univar A2000.092.1 -g n=699312 null_cells=748326 cells=1447638 min=582 max=4169 range=3587 mean=1505.60636025122 mean_of_abs=1505.60636025122 stddev=347.104721534699 variance=120481.687711681 coeff_var=23.0541481956002 sum=1052888595
- Пронормируем:
eval `r.univar A2000.092.1 -g` r.mapcalc "norm_A2000.092.1 = (A2000.092.1 - $mean)/$stddev"
- Посмотрим на результат:
r.info norm_A2000.092.1 +----------------------------------------------------------------------------+ | Layer: norm_A2000.092.1 Date: Thu Jan 6 17:22:46 2011 | | Mapset: PERMANENT Login of Creator: dima | | Location: fires | | DataBase: /home/dima/laboro/GRASSDATA | | Title: ( norm_A2000.092.1 ) | | Timestamp: none | |----------------------------------------------------------------------------| | | | Type of Map: raster Number of Categories: 255 | | Data Type: DCELL | | Rows: 1333 | | Columns: 1086 | | Total Cells: 1447638 | | Projection: Albers Equal Area | | N: 4755751.76759771 S: 4422409.14242904 Res: 250.06948625 | | E: 8747582.56252927 W: 8476175.04184751 Res: 249.91484409 | | Range of data: min = -2.660887 max = 7.673170 | | | | Data Description: | | generated by r.mapcalc | | | | Comments: | | (A2000.092.1 - 1505.6064) / 347.10472 | | | +----------------------------------------------------------------------------+
Аналогично поступим с остальными снимками:
eval `r.univar A2000.092.2 -g` r.mapcalc "norm_A2000.092.2 = (A2000.092.2 - $mean)/$stddev" eval `r.univar A2000.192.1 -g` r.mapcalc "norm_A2000.192.1 = 1/(1 + exp(- (A2000.192.1 - $mean)/$stddev))" eval `r.univar A2000.192.2 -g` r.mapcalc "norm_A2000.192.2 = 1/(1 + exp(- (A2000.192.2 - $mean)/$stddev))"
В качестве примера ниже приводится гистограмма для нормированного второго канала (12-е июля):
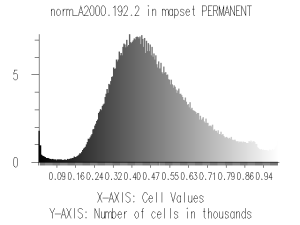 Гистограмма нормированного снимка
Гистограмма нормированного снимка
Построение нейронной сети
Загружаем R из-под GRASS GIS и считываем данные (дальнейшие команды вводятся в сеансе R):
library(spgrass6)
in1 = readRAST6('norm_A2000.092.1')
in2 = readRAST6('norm_A2000.092.2')
out1 = readRAST6('norm_A2000.192.1')
out2 = readRAST6('norm_A2000.192.2')
Создадим data.frame, содержащий считанные данные, и почистим его от пустых значений:
d = data.frame(in1 = as.vector(as.matrix(in1)), in2 = as.vector(as.matrix(in2)), out1 = as.vector(as.matrix(out1)), out2 = as.vector(as.matrix(out2))) d=d[!is.na(d$in1),] d=d[!is.na(d$out1),]
Разобъем данные на обучающее и валидационное множества, в обучающее множество поместим приблизительно 2/3 примеров:
len = length(d[,1]) val.ind = sample(1:len, size = len/3) val = d[val.ind,] train_data = d[-val.ind,]
Подгрузим AMORE - пакет для работы с нейронными сетями. (В R есть несколько пакетов, позволяющих создавать многослойные сети, пакет AMORE был выбран, в первую очередь, из-за скорости работы).
library(AMORE)
Создадим нейронную сеть. Поскольку данных для обучения имеется достаточное количество (около 700 тыс. пикселей), то и количество нейронов можно взять относительно большим. Для начала создадим сеть достаточно больших размеров - с двумя скрытыми слоями по 20 и 10 нейронов в каждом(2-20-10-2); функцию активации для всех нейронов выберем сигмоидальную (логистическую):
net = newff(n.neurons=c(2,20,10,2), learning.rate.global=0.1, momentum.global=0.01,error.criterium="LMS", hidden.layer="sigmoid", output.layer="sigmoid", method="ADAPTgdwm")
Обучим сеть в течении 500 итераций, для этого передадим процедуре обучающее и валидационное множества. На обучающем множестве будут настраиваться веса сети, валидационное использоваться для раннего останова. Полученную сеть назовем net1
result = train(net, P=train_data[,1:2], T=train_data[,3:4], Pval=val[,1:2], Tval=val[,3:4], error.criterium="LMS", report=TRUE, show.step=1, n.shows=500) net1 = result$net
В ходе экспериментов наиболее удачной сетью оказалась сеть с одним скрытым слоем и пятью нейронами в нем. В результате обучения этой сети была достигнута ошибка на обучающем множестве: 0.0188, на валидационном: 0.0190.
После обучения сети произведем расчеты, подавая в обученную сеть данные первого снимка. Для этого предварительно входные данные поместим в ненужную уже переменную d, затем в переменную y поместим результаты работы сети:
d = data.frame(in1 = as.vector(as.matrix(in1)), in2 = as.vector(as.matrix(in2)) ) y = sim(net1, d)
И, наконец, последний этап, который нужно проделать в R - экспорт результатов обратно в GRASS:
r1 = out1 r1$norm_A2000.192.1 = y[,1] writeRAST6(r1, 'net.1') r2 = out2 r2$norm_A2000.192.2 = y[,2] writeRAST6(r2, 'net.2')
Анализ результатов
После экспорта полученных растров в GRASS выходим из R. Прежде, чем сравнивать созданные нейронной сетью растры с реальными снимками, их нужно денормировать. Понятно, что денормировать нужно используя те же параметры среднего и стандартного отклонения, которые использовались при нормировании:
x = m - s*log(1/X - 1)
Для этого:
eval `r.univar A2000.192.2 -g` r.mapcalc "net_A2000.192.2 = $mean - $stddev * log(1/net.2 - 1)" eval `r.univar A2000.192.1 -g` r.mapcalc "net_A2000.192.1 = $mean - $stddev * log(1/net.1 - 1)"
В итоге получаем два растра net_A2000.192.1 и net_A2000.192.2, в которых содержатся значения для первого и второго канала соответственно, предсказанные нейронной сетью. Вместо того, чтобы сравнивать непосредственные значения полученных растров и снимков, будет удобно найти разности между предсказанным значением и реальным снимком:
r.mapcalc "razn2 = net_A2000.192.2 - A2000.192.2" r.mapcalc "razn1 = net_A2000.192.1 - A2000.192.1"
Покрасим разности для удобства восприятия, используя их статистические характеристики:
r.colors.stddev razn2 r.colors.stddev razn1
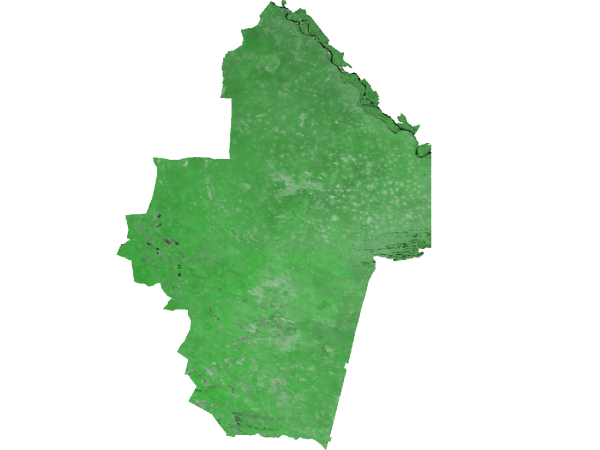
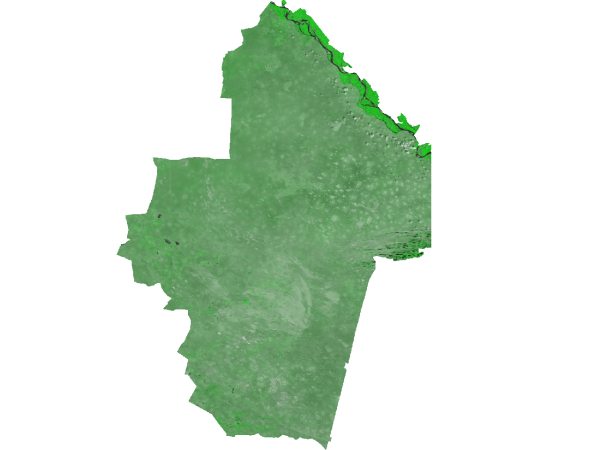
Фрагменты полученных результатов приводятся на рисунках ниже. Сначала рассмотрим исходные снимки (северо-восточный фрагмент участка):
 Долина Волги, 3-е апреля (rgb: 1-2-1)
Долина Волги, 3-е апреля (rgb: 1-2-1)
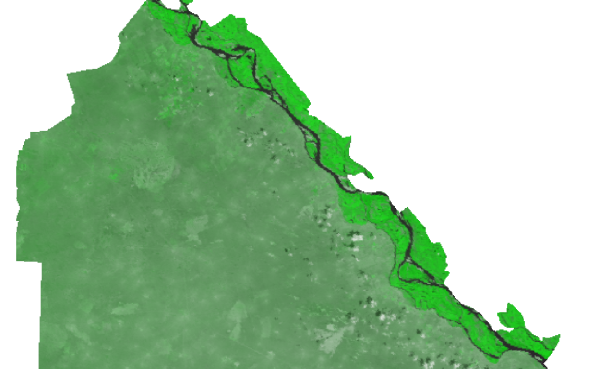 Долина Волги, 12-е июля (rgb: 1-2-1)
Долина Волги, 12-е июля (rgb: 1-2-1)
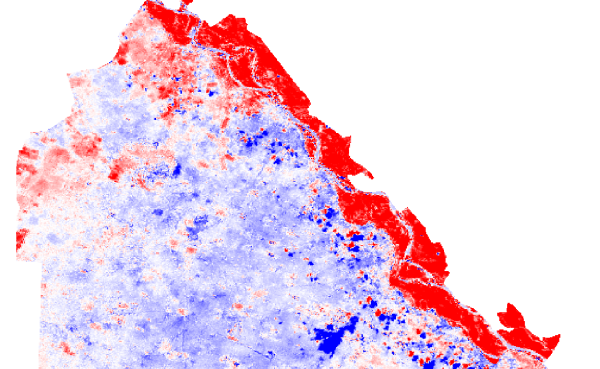
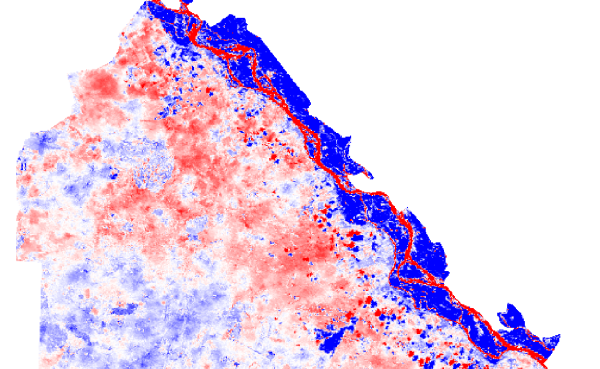
На снимках хорошо видно изменение растительности в долине Волги. Кроме того на позднем снимке заметна небольшая облачность. Степень различия предсказанных и реальных значений видна на растрах razn1 и razn2. Яркие участки соответствуют большим по модулю значениям разности, бледные участки - небольшим:
Для удобства восприятия можно объединить две разности в один растр. Первое, что приходит в голову - перемножить их, тогда большие отклонения разностей, присутствующие на обоих растрах увеличатся еще больше, а малые - уменьшатся:
r.mapcalc "razn = razn1 * razn2" r.colors.stddev razn
На полученном таким образом объединенном снимке более четко видны границы изменений: растительность в долине Волги, а также облака, появившиеся на втором снимке.
Обсудить в форуме Комментариев — 2
Ссылки по теме
Последнее обновление: September 09 2021
Дата создания: 09.01.2011
Автор(ы): Дмитрий Колесов, Максим Дубинин
© GIS-Lab и авторы, 2002-2021. При использовании материалов сайта, ссылка на GIS-Lab и авторов обязательна. Содержание материалов - ответственность авторов. (подробнее).